当前位置:网站首页>【语义分割】2015-UNet MICCAI
【语义分割】2015-UNet MICCAI
2022-08-10 19:12:00 【說詤榢】
【语义分割】2015-UNet MICCAI
网络于2015年5月提出,在后续图像分割领域广泛运用。
论文题目:U-Net: Convolutional Networks for Biomedical Image Segmentation
论文链接:https://arxiv.org/abs/1505.04597
论文代码:https://github.com/milesial/Pytorch-UNet
论文翻译:https://jokerak.blog.csdn.net/article/details/124069561
发表时间:2015年5月
引用:Ronneberger O, Fischer P, Brox T. U-net: Convolutional networks for biomedical image segmentation[C]//International Conference on Medical image computing and computer-assisted intervention. Springer, Cham, 2015: 234-241.
引用数:46490
论文分为5章
- 介绍
- 网络结构
- 训练
- 实验
- 结论
1. 简介
UNet最早发表在2015的MICCAI上,短短3年,引用量目前已经达到了4070,足以见得其影响力。
而后成为大多做医疗影像语义分割任务的baseline,也启发了大量研究者去思考U型语义分割网络。而如今在自然影像理解方面,也有越来越多的语义分割和目标检测SOTA模型开始关注和使用U型结构,
比如语义分割Discriminative Feature Network(DFN)(CVPR2018),目标检测Feature Pyramid Networks for Object Detection(FPN)(CVPR 2017)等。
2. 网络结构(创新点)
开始时,UNet主要应用在医学图像的分割,并且快速成为大多做医学图像语义分割任务的baseline,而后其他领域的学者和专家也受其启发进行了魔改。当然,也有些同学会说,这个算法中的一些思想很多人在以前也有提出,比如下采样或是多尺度的思想,但是有一个问题,在众多思路中寻找合理的方法进行组合与重构以达到更有效的结果也是一种巨大的创新和进步。
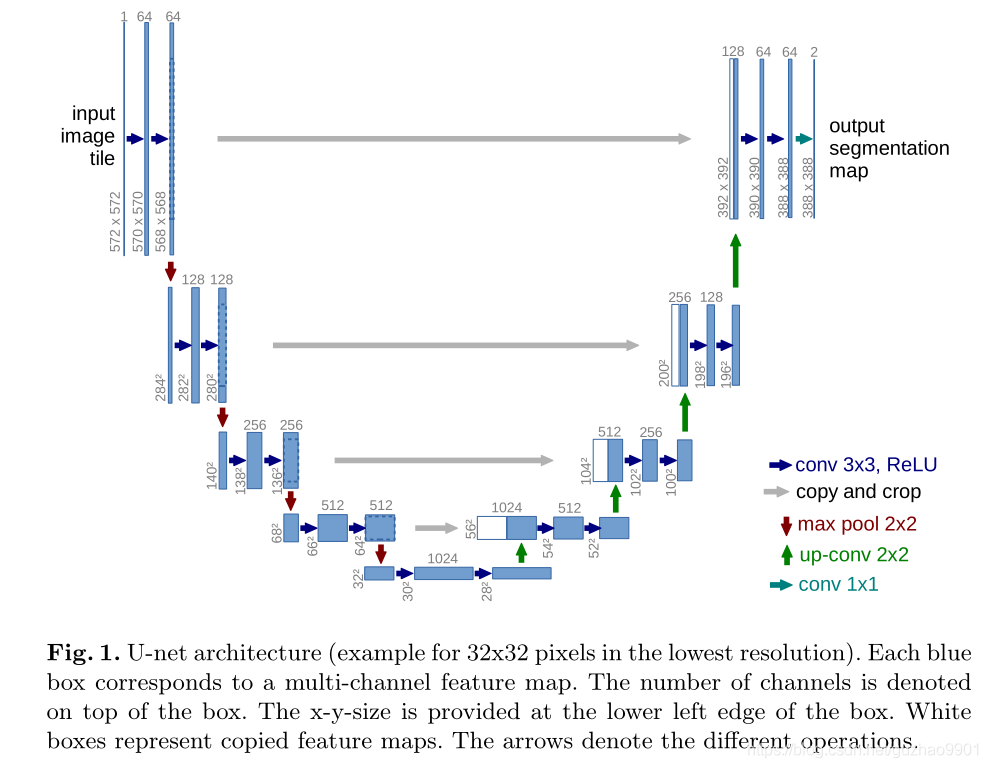
2.1 多尺度获得信息
在encoder操作中,采用了5个池化层(在实际应用中也有采用4个池化层的操作),在不断的下采样过程中不断的降低分辨率以获得不同尺度的图像信息,图像的信息由底层信息中的点、线、梯度等信息逐渐向高程信息中的轮廓以及更抽象的信息过度,整个网路完成了“由细到粗”特征的提取与组合,使UNet得到的信息更加全面。
2.2 跳跃连接
做过数字图像处理的同学们应该很清楚一点:图像从低分辨率转高分辨率图像会失真(decoder部分);而高分辨率转低分辨率则失真相对可忽略,也保留了更多的细节信息(encoder部分)!因此,在decoder中上采样过程中就失去了对细节信息的敏感。这个时候skip connection就成为真正的神来之笔,此操作将同层高度的encoder更精准的梯度、点、线等信息直接Concat到同层的decoder中,相当于在判断目标大体区域内添加细节信息,这种操作当然可以使UNet获得更准确的分割结果。
3. 代码
import torch.nn as nn
import torch
import torch.nn.functional as F
class double_conv(nn.Module):
""" UNet,VGG中的双卷积操作 """
def __init__(self, in_channels, out_channels, mid_channels=None):
""" Args: in_channels: 输入通道 out_channels: 输出通道 mid_channels: 中间通道 """
super(double_conv, self).__init__()
if not mid_channels:
mid_channels = out_channels
self.double_conv = nn.Sequential(
nn.Conv2d(in_channels, mid_channels, kernel_size=(3, 3), padding=1, bias=False),
nn.BatchNorm2d(mid_channels),
nn.ReLU(inplace=True),
nn.Conv2d(mid_channels, out_channels, kernel_size=(3, 3), padding=1, bias=False),
nn.BatchNorm2d(out_channels),
nn.ReLU(inplace=True)
)
def forward(self, x):
return self.double_conv(x)
class up(nn.Module):
""" 上采样模块 """
def __init__(self, in_channels, out_channels, bilinear=True):
super(up, self).__init__()
# 如果是双线性的,使用正规卷积来减少信道的数量
if bilinear:
self.up = nn.Upsample(scale_factor=2, mode='bilinear', align_corners=True)
self.conv = double_conv(in_channels, out_channels, in_channels // 2)
else:
self.up = nn.ConvTranspose2d(in_channels, in_channels // 2, kernel_size=(2, 2), stride=(2, 2))
self.conv = double_conv(in_channels, out_channels)
def forward(self, x1, x2):
# 1. 第一步对x1做上采样。然后和x_2进行拼接
x1 = self.up(x1)
diffY = x2.size()[2] - x1.size()[2]
diffX = x2.size()[3] - x1.size()[3]
# 这里是怕上采样完毕的x1维度和x_2有所差别
x1 = F.pad(x1, [diffX // 2, diffX - diffX // 2,
diffY // 2, diffY - diffY // 2])
# 在通道上进行拼接,所以维度为1
x = torch.cat([x2, x1], dim=1)
return self.conv(x)
class down(nn.Module):
""" 用maxpool降尺度,然后用double conv """
def __init__(self, in_channels, out_channels, ):
super(down, self).__init__()
self.down = nn.Sequential(
nn.MaxPool2d(2),
double_conv(in_channels, out_channels)
)
def forward(self, x):
return self.down(x)
class OutConv(nn.Module):
def __init__(self, in_channels, out_channels):
super(OutConv, self).__init__()
self.co = nn.Sequential(
nn.Conv2d(in_channels, out_channels, kernel_size=(1, 1)),
)
def forward(self, x):
return self.co(x)
class unet(nn.Module):
def __init__(self, num_classes, bilinear=False):
super(unet, self).__init__()
# unet 一共4次下采样,4次上采样
# 第一步,首先来个双卷积
self.conv1 = double_conv(3, 64)
# 再来4步下采样
self.down1 = down(64, 128)
self.down2 = down(128, 256)
self.down3 = down(256, 512)
""" 如果使用了转置卷积的话,factor=1.因为可以使用转置卷积让通道数变成原来的1/2 如果使用了线性差值,factor=2.这样子的话。第四层的通道数就不需要升级通道 """
factor = 2 if bilinear else 1
self.down4 = down(512, 1024 // factor)
self.up1 = up(1024, 512 // factor, bilinear)
self.up2 = up(512, 256 // factor, bilinear)
self.up3 = up(256, 128 // factor, bilinear)
self.up4 = up(128, 64, bilinear)
self.outc = OutConv(64, num_classes)
def forward(self, x):
x1 = self.conv1(x)
x2 = self.down1(x1)
x3 = self.down2(x2)
x4 = self.down3(x3)
x5 = self.down4(x4)
x = self.up1(x5, x4)
x = self.up2(x, x3)
x = self.up3(x, x2)
x = self.up4(x, x1)
return self.outc(x)
if __name__ == '__main__':
x=torch.randn(1,3,224,224)
model=unet(num_classes=19)
y=model(x)
print(y.shape)
4. 问题
4.1 为啥医学图像在UNet中有明显的性能提升
我感觉,最重要的因素是由于医学图像的特殊性决定的。做过B超、核磁、CT的同学看到生成的图像的直观感觉是什么?首先,图像并没有显示自然图像那样清晰,边界很模糊,对比度差;其次,噪声点相对较多,不同个体呈现出的噪声信号也不稳定;最后,医学图像的信号复杂度并不低,人体不同组织成像是完全不同的,而且绝大部分图像都是灰度图像(绝大部分彩色是伪彩,细胞类除外),灰度范围也较大。
而以上医学图像特点就注定了UNet在此领域的广泛应用。首先,多尺度的信息提取,细节与“较粗”抽象信息都得到有效的提取与保留,在降低了噪声的影响的同时最大限度的保留模糊边界的梯度信息。最后,医学图像虽然信号复杂但是类别并不复杂,且人体组织分布有一定的规则(哪怕有一定的病变毕竟都是同一个祖先进化来的),语义分割相对也简单。
当然,医学图像的数据量也是重要因素,但是小样本的网络也挺多的,迁徙学习网络的泛化能力也挺强的,我感觉医学图像使用UNet最重要的是以上两点。
参考链接
Unet论文详解U-Net:Convolutional Networks for Biomedical Image Segmentation_祥瑞Coding的博客-CSDN博客_unet论文
图像分割必备知识点 | Unet详解 理论+ 代码 - 知乎 (zhihu.com)
图像分割UNet系列------UNet详解_gz7seven的博客-CSDN博客_unet结构图
原文链接:https://blog.csdn.net/guzhao9901/article/details/119461005
边栏推荐
- (12) findContours function hierarchy explanation
- 2022 Hangdian Multi-School Seven Black Magic (Sign-in)
- flask的配置文件
- 关于npm/cnpm/npx/pnpm与yarn
- Tf铁蛋白颗粒包载顺铂/奥沙利铂/阿霉素/甲氨蝶呤MTX/紫杉醇PTX等药物
- 常用Anaconda安装错误解决办法Traceback (most recent call last):[通俗易懂]
- [SemiDrive source code analysis] [MailBox inter-core communication] 52 - DCF Notify implementation principle analysis and code combat
- whois information collection & corporate filing information
- 电脑为什么会蓝屏的原因
- mysql----group by、where以及聚合函数需要注意事项
猜你喜欢

『牛客|每日一题』岛屿数量

端口探测详解
whois信息收集&企业备案信息
测试/开发程序员值这么多钱么?“我“不会愿赌服输......
QoS服务质量六路由器拥塞管理
电脑重装系统Win11格式化硬盘的详细方法
YOLOv3 SPP源码分析
WCF and TCP message communication practice, c # 】 【 realize group chat function
Transferrin-modified vincristine-tetrandrine liposomes | transferrin-modified co-loaded paclitaxel and genistein liposomes (reagents)
铁蛋白颗粒负载雷替曲塞/培美曲塞/磺胺地索辛/金刚烷(科研试剂)
随机推荐
代理模式的使用总结
3D Game Modeling Learning Route
Hangdian Multi-School Seven 1003-Counting Stickmen (Combination Mathematics)
电脑如何去掉u盘写保护的状态
Metasploit——渗透攻击模块(Exploit)
常见端口及服务
【知识分享】在音视频开发领域中SEI到底是个啥?
铁蛋白颗粒Tf包载多肽/凝集素/细胞色素C/超氧化物歧化酶/多柔比星(定制服务)
巧用RoaringBitMap处理海量数据内存diff问题
GBASE 8s 高可用RSS集群搭建
Redis persistence mechanism
(12) findContours function hierarchy explanation
30分钟使用百度EasyDL实现健康码/行程码智能识别
Optimization is a habit The starting point is to 'stand close to the critical'
Echart饼状图标注遮盖解决方案汇总
QoS服务质量七交换机拥塞管理
mysql----group by、where以及聚合函数需要注意事项
argparse——命令行参数解析
转铁蛋白修饰蛇床子素长循环脂质体/负载三七皂苷R1的PEG-PLGA纳米粒([email protected] NPs)
QoS Quality of Service Seven Switch Congestion Management