当前位置:网站首页>基于GPU实例的Nanopore数据预处理
基于GPU实例的Nanopore数据预处理
2022-04-23 16:07:00 【4BasesTeam】
说明
本文为Nanopore碱基识别及质控简明教程,正文将使用到如下软硬件:
- GPU计算型GN7 | GN7.5XLARGE80实例:腾讯云提供的实例,本教程利用该实例搭建测试环境(本测试使用的CentOS 7.5 64位操作系统)。
- NVIDIA Tesla 驱动:显卡驱动是硬件与系统沟通的软件配套。
- CUDA计算框架:NVIDIA 推出的只能用于自家GPU的并行计算框架。
- Guppy软件:Nanopore官方提供的碱基识别软件。
- MinIONQC脚本:质控脚本。
Nanopore测序背景
测序原理
纳米孔是一个纳米级的小孔,在其设备中,Oxford Nanopore 使离子电流通过纳米孔,并测量当生物分子通过或靠近纳米孔时的电流变化。由于纳米孔的直径非常细小,仅允许单个核酸聚合物通过,而ATCG单个碱基的带电性质不一样,因此不同碱基通过蛋白纳米孔时对电流产生的干扰不同,通过实时监测并解码这些电流信号便可确定碱基序列,从而实现测序。
图片来源:Oxford Nanopore Technologies官网
Nanopore优势
- 超长读长:MinION用户迄今为止报告的最长读取长度为>4 Mb。
- 便携快捷:设备轻巧,文库制备快速。
- 直接测序:不同于Illumina及Pacbio的光学测序系统,Nanopore是基于电学信号的检测,可省去扩增,规避了扩增偏好性的风险,直接读取DNA/RNA分子电信号来分析碱基类型,亦提供了表观遗传学分析的机会。
- 实时测序:与在运行结束时批量交付数据的传统测序技术不同,纳米孔技术提供的是动态、实时的测序。
前置驱动及软件安装
GPU Tesla驱动安装
1. 底层模块检查:
rpm -qa | grep -i dkms
rpm -qa | grep kernel-devel
rpm -qa | grep gcc如未能找到,则参考如下命令安装:
yum install -y dkms
yum install -y kernel-devel
yum install -y gcc
#或yum install -y dkms kernel-devel gcc2. 下载驱动:
wget https://us.download.nvidia.com/tesla/510.47.03/NVIDIA-Linux-x86\_64-510.47.03.run细节可参考:https://cloud.tencent.com/document/product/560/80483. 安装驱动:
chmod +x NVIDIA-Linux-x86_64-418.126.02.runsh NVIDIA-Linux-x86_64-418.126.02.run4. 验证:
nvidia-smi
#监控GPU使用情况如返回信息类似下图中的 GPU 信息,则说明驱动安装成功。
CUDA框架部署
1. 框架下载
wget https://developer.download.nvidia.com/compute/cuda/11.6.2/local_installers/cuda-repo-rhel7-11-6-local-11.6.2_510.47.03-1.x86_64.rpm细节可参考:https://cloud.tencent.com/document/product/560/8064
2. 安装框架
rpm -i cuda-repo-rhel7-11-6-local-11.6.2_510.47.03-1.x86_64.rpm
yum clean all
yum -y install nvidia-driver-latest-dkms cuda
yum -y install cuda-driversGuppy部署
1. Guppy下载
wget https://mirror.oxfordnanoportal.com/software/analysis/ont-guppy_6.0.1_linux64.tar.gz2. Guppy解压及配置
tar xf ont-guppy\_6.0.1\_linux64.tar.gz
#可直接配置到工作环境环境中~/.bashrc
#本示例解压在/home目录下,可自行替换实际软件路径
#export PATH="/home/ont-guppy/bin:$PATH"MinIONQC部署
1. 前置R及R包部署
yum install R
Rscript -e "install.packages(c('ggplot2','viridis','plyr','reshape2','readr','yaml','scales','futile.logger','data.table','optparse'), repos='https://mirrors.tuna.tsinghua.edu.cn/CRAN/')"2. 下载R脚本
wget https://raw.githubusercontent.com/roblanf/minion_qc/master/MinIONQC.R -O MinIONQC.R数据准备
本次的测试数据来源于NCBI的PRJNA812612项目的开放数据集,github上有作者的model JSON文件及分析脚本,感兴趣的可以自行前往(https://github.com/DamLabResources/HIV-Quasipore-basecallers)。
1. 下载地址:
2. 数据下载
wget https://sra-pub-src-2.s3.amazonaws.com/SRR18215551/JLat_106_MinION_R941.fast5.tar.gz.2
#AWS直接下载的速度较快腾讯云的下载速度也比较给力,如下图
3. 数据解压
tar xf JLat_106_MinION_R941.fast5.tar.gz.2 碱基识别
Guppy核心参数说明
|
参数 |
说明 |
本次所用值 |
|---|---|---|
|
-i |
Path to input fast5 files. |
/home/nanopore/data/r9.4.1/ |
|
-s |
Path to save fastq files. |
/home/nanopore/normal/r9.4.1 |
|
-c |
Config file to use. |
dna_r9.4.1_450bps_hac.cfg |
|
-x |
Specify basecalling device: 'auto', or 'cuda:<device_id>'. |
cuda:0 |
关于config参数,大家可以通过如下命令(列出Guppy支持的试剂盒和芯片清单),增加理解。
guppy_basecaller --print_workflowsBasecalling命令
guppy_basecaller -s /home/nanopore/normal/r9.4.1 -i /home/nanopore/data/r9.4.1/ -x cuda:0 -c dna_r9.4.1_450bps_hac.cfg 感兴趣的可以查看GPU效能,这里可以看到用了8G的显存,显存核心利用率为100%,具体参数可以参考网上博客说明,如https://www.jianshu.com/p/ceb3c020e06b?msclkid=4dc4c38ac2c311ec99ef9b6bf6e3371a
本次Basecalling总共用了近58分钟,T4的速度比V100慢3~4倍,如果是V100,这个速度一般在20分钟以内。
本次生成70个fastq文件,大家在使用时候可以直接合并成一个文件。
cat *.fastq > AllinOne.fastq数据质控
核心参数说明
|
参数 |
说明 |
本次所用值 |
|---|---|---|
|
-i |
Input file or directory (required) |
/home/nanopore/normal/r9.4.1 |
|
-q |
The cutoff value for the mean Q score of a read (default 7) |
12 |
|
-o |
Output directory (optional, default is the same as the input directory). |
/home/nanopore/normal/r9.4.1/qc |
|
-p |
Number of processors to use for the anlaysis (default 1) |
20 |
质控命令
Rscript MinIONQC.R -i /home/nanopore/normal/r9.4.1 -q 12 -o /home/nanopore/normal/r9.4.1/qc -p 20QC完成后,将生成一些质控图及统计文件,感兴趣的小伙伴可以到github上(https://github.com/roblanf/minion_qc
)看详细的解释说明,这里就不展开说明了。
后续的工作,大家可以根据各自需求调整或进行下游二三级分析。
参考资料
- https://nanoporetech.net/how_it_works
- https://nanoporetech.com/how-it-works/advantages-nanopore-sequencing
- https://zhuanlan.zhihu.com/p/91960495
- https://cloud.tencent.com/document/product/560/8048
- https://cloud.tencent.com/document/product/560/8064
- https://www.ncbi.nlm.nih.gov/bioproject/812612
- https://www.jianshu.com/p/ceb3c020e06b?msclkid=4dc4c38ac2c311ec99ef9b6bf6e3371a
- https://github.com/roblanf/minion_qc
版权声明
本文为[4BasesTeam]所创,转载请带上原文链接,感谢
https://cloud.tencent.com/developer/article/1986121
边栏推荐
- Method 2 of drawing ROC curve in R language: proc package
- Spark 算子之distinct使用
- Homewbrew installation, common commands and installation path
- 面试题 17.10. 主要元素
- Spark 算子之coalesce与repartition
- Questions about disaster recovery? Click here
- PS为图片添加纹理
- Config组件学习笔记
- Master vscode remote GDB debugging
- Application case of GPS Beidou high precision satellite time synchronization system
猜你喜欢
R语言中实现作图对象排列的函数总结
Install redis and deploy redis high availability cluster
Config learning notes component
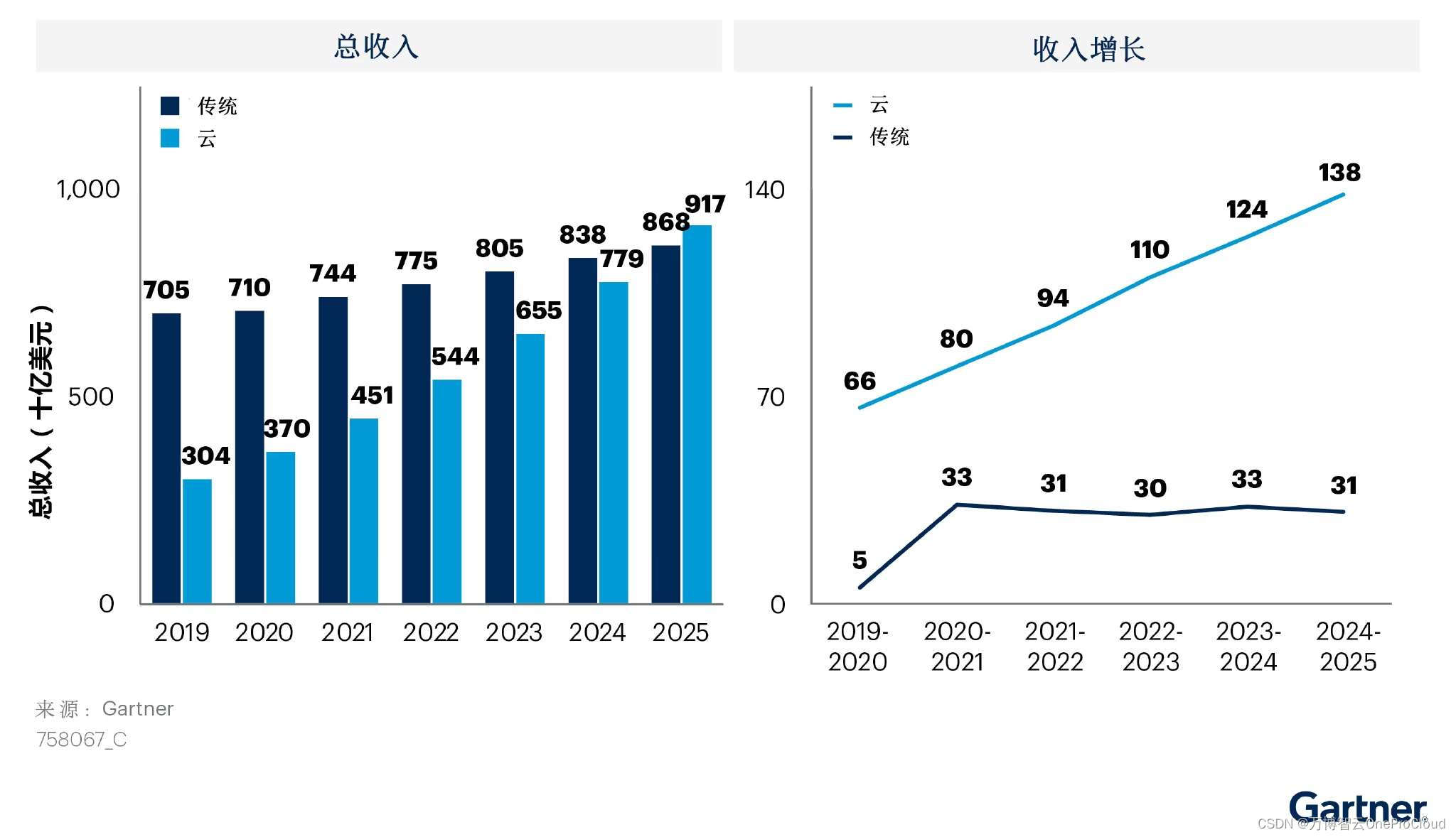
Gartner predicts that the scale of cloud migration will increase significantly; What are the advantages of cloud migration?
Spark 算子之distinct使用
C language self compiled string processing function - string segmentation, string filling, etc

Best practice of cloud migration in education industry: Haiyun Jiexun uses hypermotion cloud migration products to implement progressive migration for a university in Beijing, with a success rate of 1
Spark 算子之groupBy使用

Review 2021: how to help customers clear the obstacles in the last mile of going to the cloud?
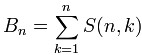
C#,贝尔数(Bell Number)的计算方法与源程序
随机推荐
5 minutes, turn your excel into an online database, the magic cube net table Excel database
捡起MATLAB的第(7)天
ESP32_ Arduino
一文掌握vscode远程gdb调试
[AI weekly] NVIDIA designs chips with AI; The imperfect transformer needs to overcome the theoretical defect of self attention
299. Number guessing game
Meaning and usage of volatile
Win11/10家庭版禁用Edge的inprivate浏览功能
Upgrade MySQL 5.1 to 5.610
[split of recursive number] n points K, split of limited range
Day (10) of picking up matlab
Simple usage of dlopen / dlsym / dlclose
[key points of final review of modern electronic assembly]
shell_ two
Partitionby of spark operator
JVM - Chapter 2 - class loader subsystem
gps北斗高精度卫星时间同步系统应用案例
Research and Practice on business system migration of a government cloud project
Hyperbdr cloud disaster recovery v3 Release of version 3.0 | upgrade of disaster recovery function and optimization of resource group management function
JSP learning 3